Prof. Dr. Wolfgang Streit
Die zentralen Forschungsaspekte der AG Streit
Für Interessierte sind hier einige Medienbeiträge, die Ausschnitte aus unserer Forschung zeigen:
- DAS! (NDR 06.09.2024) „Plastikfressende Bakterien: Enzym soll PET zersetzen“
- Hamburg Journal (NDR 16.10.2023) „Forscher entdecken Enzym zur Zersetzung von Plastikflaschen“ Video
- Tagesschau (NDR 07.10.2023) „Forschung zum Abbau von Mikroplastik“
- Schleswig-Holstein Magazin (NDR 29.09.2023) „Plastik-zersetzende Bakterien in der Tiefsee“ Video
- Leschs Kosmos (ZDF 16.07.2019) „Die Plastik-Zeitbombe: Wege aus der Krise“
- Biokatalysatoren für die Biotechnologie, vorgestellt von Prof. Dr. Wolfgang Streit (2019) Video
Außerdem:
Schauen Sie mit einem Klick in die PAZy-Datenbank, die ausschließlich biochemisch charakterisierte kunststoffaktive Enzyme auflistet – kuratiert in Zusammenarbeit zwischen der Universität Stuttgart, Institut für Biochemie und Technische Biochemie und der Universität Hamburg, Abteilung Mikrobiologie & Biotechnologie.
I. Metagenomik, Biotechnologie, enzymatischer Plastikabbau
In einem der zentralen Forschungsgebiete nutzen wir die Metagenomik (Genome aller Mikroorganismen), um neue Biokatalysatoren und andere wertvolle Biomoleküle für unterschiedliche Anwendungen der Biotechnologie bereit zu stellen. Ein zentraler Aspekt unserer Forschung liegt in der Nutzbarmachung der nicht-kultivierten Organismen bzw. deren Metagenomen für die Biotechnologie. Heute weiß man, dass 99 % aller Mikroorganismen nicht oder nur schwer kultivierbar sind. Dieses nahezu unbegrenzte Potential neuer Enzyme und Moleküle wollen wir nutzbar machen. Zur Nutzung dieses immensen Potentials entwickeln wir einerseits neue Technologien anderseits identifizieren wir viele Biokatalysatoren. Dies geschieht gleichermaßen vor dem Hintergrund der Grundlagen- und der angewandten Forschung.
- Biokatalysatoren aus dem Metagenom für die chemische Industrie: Mit Hinblick auf die Identifizierung und Bereitstellung von Biokatalysatoren haben wir zwar relativ unterschiedliche Enzymklassen bearbeitet, unser Schwerpunkt liegt aber auf der Isolierung von Lipasen, Esterasen und anderen Hydrolasen für industrielle Anwendungen. Diesbezüglich verfügen wir mittlerweile über ein der größten Lipase/Esterase Enzymkollektionen mit über 150 aktiven und charakterisieren Biokatalysatoren (Metacat-Kollektion) und etwa 80 aktiven cellulolytischen Enzymen. Hinzu kommen Laktonasen, Hydrogenasen, Glykosyltransferasen, Decarboxlyasen und andere Enzyme, die wir oft bis zur 3D-Struktur charakterisiert haben. Bei unseren Arbeiten sind wir auch daran interessiert komplette neue Reaktionen und Enzyme zu identifizieren (Dark und Grey Matter-Proteine). Begleitend entwickeln wir meist Reporterassays zur Detektion der Reaktionen im Hochdurchsatzverfahren. Unsere Metagenom-Enzymkollektion bauen wir stetig aus, und sie bildet die Grundlage für viele unserer Forschungsprojekte.
- Enzyme für den Abbau von synthetischen Polymeren: Im Zuge der o. a. Metagenomsuchen haben wir vor wenigen Jahren damit begonnen PET-abbauende Enzyme zu isolieren. Wir verfügen derzeit über etwa 60-PET-aktive Enzymen aus ganz unterschiedlichen Organismen, die in unseren Labors identifiziert und teilweise charakterisiert wurden. Gerade diese Enzyme stehen derzeit im Fokus von zwei BMBF-Projekten (LipoBiocat und Plastisea) und stellen weltweit die größte Kollektion solcher Enzyme dar. Diese Arbeiten erweiterten wir derzeit auch auf andere Kunststoffe, für die es bisher keine bekannten Enzyme gibt. Prinzipiell gehen wir davon aus, dass nicht nur der Abbau von Interesse ist, sondern auch die Verwendung der Enzyme zur Synthese neuer bioabbaubarer Polymere und Materialien. Diese Arbeiten bilden derzeit die Grundlage für eine Reihe von laufenden Doktorarbeiten.
- Etablierung einer digitalen Plattform für Enzymsuchen: Eine Herausforderung bei vielen unserer Projekte, die wir oft in Zusammenarbeit mit Industriepartner durchführen, wird durch die relativ langen Laufzeiten bewirkt. Die übliche Entwicklungszeit beträgt dabei meist 3–5 Jahre, gerechnet von der Identifizierung des Enzyms bis hin zu ersten Upscaling Versuchen. Um diesen Zeitraum zu verkürzen, arbeiten wir mit Hochdruck an einer automatisierten Plattform. Wir wollen die Bereitstellung von robusten Enzymen oder ganzen Stoffwechselwegen für die Biotechnologie in relativ kurzen Zeiträumen von maximal 3–6 Monaten erreichen. Hier arbeiten wir an der Entwicklung von in vitro-Systemen, um einen wirklich schnellen Zugang zu den nicht-kultivierten Organismen zu erhalten. Ziel ist es dabei, in Kombination mit sequenzbasierter Metgenomsuche und in vitro Ansätzen einige 1000 aktive Enzyme in sehr kurzer Zeit für initiale Screening bereitzustellen und erst in einem zweiten Schritt in ein Upscaling einzusteigen. Die Plattform wird in wenigen Jahren verfügbar sein. Derzeit können wir etwa 30–50 Enzyme innerhalb sehr kurzer Zeit bereitstellen.
II. Biofilme: Analytik, Architektur und Omics
Mikrobielle Biofilme sind oft unerwünschte Aufwüchse auf Oberflächen und sie stellen sowohl im klinischen als auch im industriellen Umfeld eine echte Herausforderung dar.
- Biofilme – Detektion und Analytik: Die Biofilmanalytik und zuverlässige Diagnostik ist in der Industrie und auch im klinischen Umfeld oft sehr schwierig und aufwendig. Hierzu analysieren wir Metagenome- von komplexen Biofilmen und gewinnen so tiefe Einblicke in die Funktionsweise dieser Konsortien. Wir nutzen die ganze Bandbreite der omics-Analyse und auch Bild-darstellende Verfahren, wie die Elektronenmikroskopie, sind feste Bestandteile solcher Analysen. Auch die EPS-Analytik haben wir etabliert.
- Kontrolle und Steuerung von Wachstum an Grenzflächen: Hierzu arbeiten wir intensiv an der Aufklärung von Prozessen, die für die bakterielle Kommunikation bei der Biofilmbildung eine Rolle spielen. In diesem Forschungsgebiet interessiert uns vor allen Dingen, wie Mikroorganismen miteinander reden. Hier sind wir einerseits auf der Suche nach neuen Signalmolekülen, anderseits gehen wir auch der Frage nach, wie sich Wachstum in einem Biofilm gezielt steuern lässt. Dazu arbeiten wir sowohl mit gemischten Konsortien auf der Ebene von Gesamtpopulationen als auch im Bereich der Einzelzellanalyse. Bei diesen Arbeiten rekonstruieren wir zudem Multispeziesbiofilme und versuchen dabei die natürlichen Bedingungen im Prozess oder klinischen Umfeld nachzustellen. Auf diese Weise erhalten wir sehr tiefe Einblick in die 3D-Biofilmstrukturen und Omics.
- Vermeidung mikrobieller Biofilme: Prinzipiell ist es besser, Biofilme sehr früh im Wachstum zu inhibieren. Hier arbeiten wir zusammen mit den Kollegen aus der Chemie sowohl an enzymatischen als auch an Compound-getriebenen Ansätzen, um mikrobielle Biofilme zu inhibieren. Dabei haben wir erste Erfolge mit Enzymen erzielen können, die in die Zell-Zellkommunikation eingreifen, aber auch mit Molekülen aus der Stoffgruppe der Flavonoide und Diorcinole. Das langfristige Ziel dieser Arbeiten ist es die Biofilmpopulation so zu verändern, dass sich keine (kaum) Biofilme bilden, oder aber eine rasche Ablösung stattfindet.
III. Mikrobielle Diversität und ‚One Health‘
Die zunehmende Freisetzung von Antibiotika und anderen Schadstoffen wirkt sich direkt und indirekt auf die mikrobielle Vielfalt aus und fördert die Ausbreitung bakterieller Resistenzmechanismen, die ein hohes Risiko für die Gesundheit von Mensch und Tier darstellen. Darüber hinaus beschleunigen Klimawandel und andere humane Faktoren diese Auswirkungen. Es gibt keine klaren Methoden und Modelle, die die Auswirkungen dieser Faktoren auf die biologische Vielfalt und die menschliche Gesundheit in städtischen Regionen bewerten.
Deshalb soll das Projekt MOMOBIO2.0 detaillierte, hauptsächlich omik-basierte Datensätze und Modelle entwickeln, um die urbane Mikrobiota und Veränderungen von Biodiversität und Resistenzprofil im urbanen Wasserzyklus zu verfolgen, zu überwachen und vorherzusagen. Hauptziel ist es, Veränderungen in der mikrobiellen Biodiversität als indirektes Maß für die Gesundheit von Mensch und Tier zu nutzen (One Health Konzept). Wir etablieren genetische Marker, um Veränderungen in Biodiversität und Resistenz innerhalb der Abwasserströme und den Oberflächengewässern zu verfolgen.
Dieses Netzwerkprojekt wird in enger Zusammenarbeit mit allen relevanten Partnern in Bezug auf Abwasser und Wasserströme in Hamburg durchgeführt, darunter das Universitätsklinikum Hamburg-Eppendorf (UKE), Hamburg Wasser (HW), Forschungsteams der Universität Hamburg (UHH), die lokalen Behörden (HU), die NGO Life Science Nord (LSN) und Daten aus der Hamburg City Health Study (HCHS).
Die in dem Projekt gewonnenen Daten und die neu entwickelten Modellierungen sollen es Entscheidungsträgern in Politik und Wirtschaft erleichtern geeignete Maßnahmen zur Aufrechterhaltung der Wasserqualität zu treffen und damit zur Gesunderhaltung von Mensch und Tier beizutragen. Der Projektpartner UKE trägt mit seiner Expertise in kulturellen Hochdurchsatzverfahren (culturomics), quantitativer PCR und bioinformatischer Analyse komplexer Genom- und Metagenomdaten zum Projekt bei.
Vorhandenes Methodenspektrum
Methodisch ist die Gruppe aufgrund der Themenvielfalt sehr breit aufgestellt und verfügt über viel Expertise im Bereich der DNA- und RNA-Technologien, Genomik und Metagenomik, Transkriptomik, GC-, HPLC-Analytik sowie der Proteinaufreinigung. Zudem verfügt das Labor über Expertise im Umgang mit einer Reihe von Modellorganismen, die gerade in der Biotechnologie und industriellen Mikrobiologie von Bedeutung sind. Dazu zählen z. B. Pseudomonas sp., Burkholderia sp. und andere. Im Rahmen der biotechnologischen Arbeiten entwickeln wir zudem Mikroorganismen zur Produktion. Für alle Organismen haben wir genetische Systeme etabliert. Letztendlich haben wir gerade in den letzten Jahren damit begonnen, die Einzelzelltechnologie im Labor zu etablieren und haben dazu ein erstes Lasertrapping-System etabliert. Zudem verfügen wir über 3D-Biofilm-Systeme und ein konfokales Laserscanning System zur Biofilmanalytik. Darüber hinaus entwickeln wir oft neue Techniken für den Nachweis von Enzymaktivitäten in den Metagenombanken.
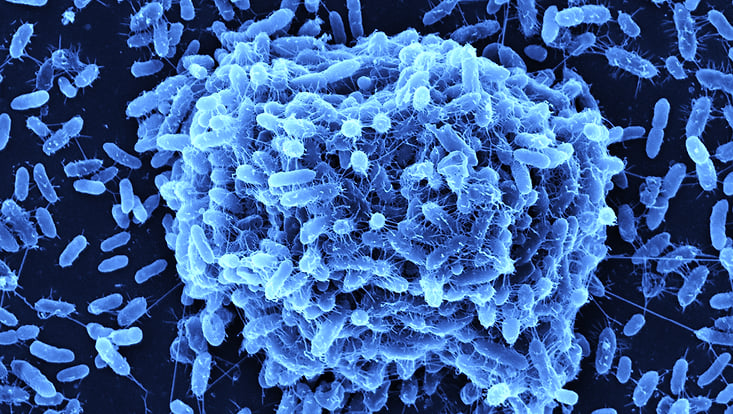
Foto: UHH/Mikrobiologie
Bakterielle Krankheitserreger können auf Wirtsgeweben wie den Atemwegen und der Haut sowie auf abiotischen Oberflächen wie medizinischen Geräten und Implantaten haften und Biofilme bilden. Bei immungeschwächten Patienten besteht aufgrund dieser Biofilme ein hohes Risiko von Krankenhausinfektionen. Unser Forschungsschwerpunkt liegt auf der Etablierung von Multispezies-Biofilmmodellen mit den folgenden Modellorganismen: Stenotrophomonas maltophilia, Pseudomonas aeruginosa, Staphylococcus aureus und dem Pilz Candida albicans. Anhand dieser Biofilmmodelle untersuchen wir die Interaktion zwischen den Spezies, die Schichtbildung, die Genexpressionsprofile mithilfe der RNA-Seq-Analyse und die Auswirkungen stammspezifischer Bakteriophagen auf die Biofilmbildung und -strukturen.
Das Bild zeigt eine Biofilmbildung des humanpathogenen Stenotrophomonas maltophilia.
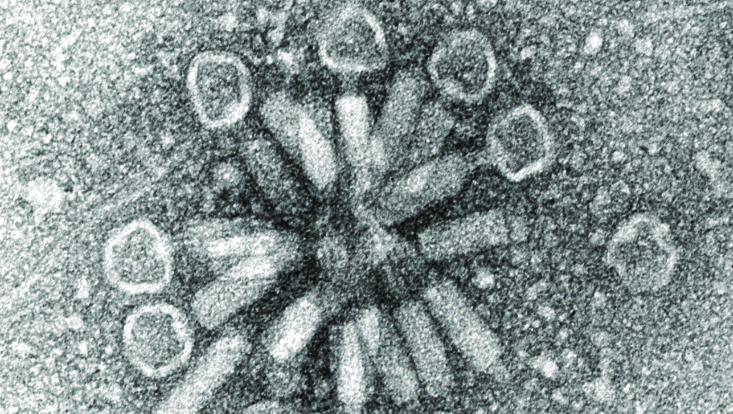
Foto: UHH/Mikrobiologie
Stenotrophomonas maltophilia phage
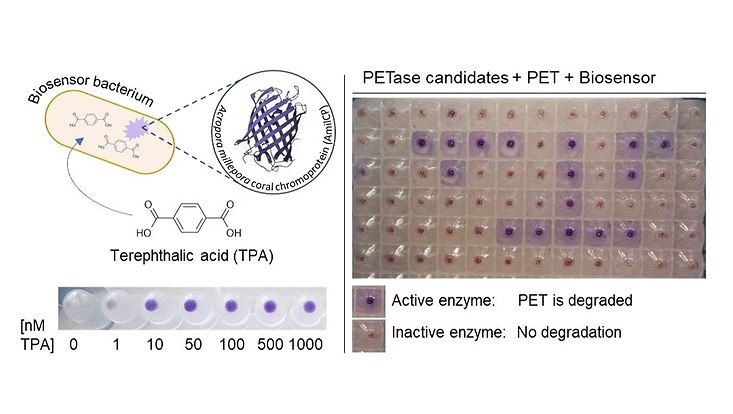
Foto: UHH/Mikrobiologie
Biosensoren für die Entdeckung neuer PET-abbauender Enzyme. Ein bakterieller Biosensor produziert ein sichtbares blaues Korallenchromoprotein, wenn er das PET-Abbauprodukt Terephthalsäure (TPA) erkennt. Empfindliche Screening-Systeme wie diese können helfen, neue PETasen mit hohen und niedrigen Aktivitäten aus vielen verschiedenen Quellen zu entdecken. (Applied and Environmental Microbiology, Vol. 89, No. 1, Dierkes et al., 2022)

Foto: UHH/Mikrobiologie
Mikroskopische Aufnahmen von fluoreszmarkierten Pathogenen im Biofilm. Die verschiedenen Spezies Stenotrophomonas maltophilia, Pseudomonas aeruginosa und Candida albicans wurden durch die Integration des gewünschten Fluoreszenzgens in das jeweilige Genom fluoreszenzmarkiert. Bei Staphylococcus aureus hingegen fand die Integration des Fluoreszenzgens extrachromosomal über das Plasmid PCM29 statt. (Genetics and Molecular Biology, Alio et al., 2023)

Foto: UHH/Mikrobiologie
Mikroskopische Aufnahmen eines Tripel-Spezies-Biofilms nach 24h, 48h und 72h. Der Biofilm besteht aus den Pathogenen Stenotrophomonas maltophilia K279a tdTomato (rot), Staphylococcus aureus SH1000 AmCyan (cyan) und Candida albicans SC5314 sfGFP (grün).
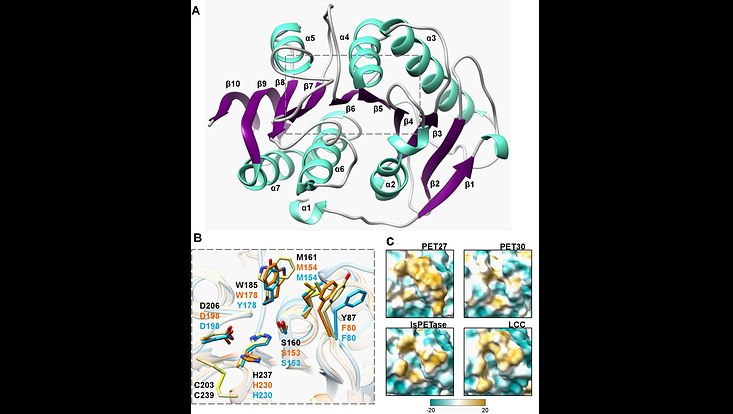
Foto: UHH/Mikrobiologie
Kristallstruktur des PET-hydrolysierenden, bacteroidetalen PET30∆PorC mit Vergleich des aktiven Zentrums und der Hydrophobizität. (A) Gesamtstruktur von PET30∆PorC. Die Struktur wurde durch Röntgenkristallographie mit einer Auflösung von 2,1 Å gelöst. (B) Vergleich der Reste des aktiven Zentrums. Alle drei Enzyme PET30∆PorC (hellblau), PET27 (orange) und IsPETase (hellgelb) haben die typischen Reste von Ser-Hydrolasen an den katalytisch aktiven Stellen (Ser, His und Asp). Die Reste von IsPETase sind in schwarzer Farbe dargestellt. (C) Oberflächenhydrophobie um den Tunnel, der zum aktiven Zentrum von vier PET-abbauenden Enzymen führt. Hydrophile Bereiche sind in Türkis und hydrophobe in Gold dargestellt. Entnommen aus Zhang et al. 2022 (Front. Microbiol. 12:803896. doi: 10.3389/fmicb.2021.803896)

Foto: Pablo Pérez-García
Plastikverschmutzter Strand auf Mallorca, Juli 2021
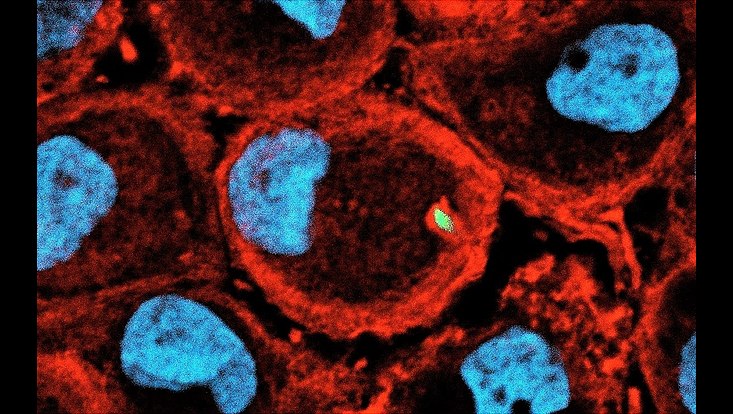
Foto: UHH/Mikrobiologie
Stenotrophomonas maltophilia ist ein opportunistischer Krankheitserreger, der von klinischer Relevanz ist. Makrophagen stellen die erste Linie der Immunabwehr bei einer bakteriellen Infektion dar. Unter Verwendung verschiedener klinischer Isolate von S. maltophilia untersuchen wir derzeit die Interaktion dieses Bakteriums mit primären menschlichen Makrophagen. Färbungen: Makrophagen F-Aktin mit Phalloidin 568 (rot), Zellkern mit DAPI (blau), S. maltophilia sind mit GFP (grün)

Foto: UHH/Mikrobiologie
Stenotrophomonas maltophilia forming very long filamentous cells and outer membrane vesicles. Image derived from Abda et al., 2015
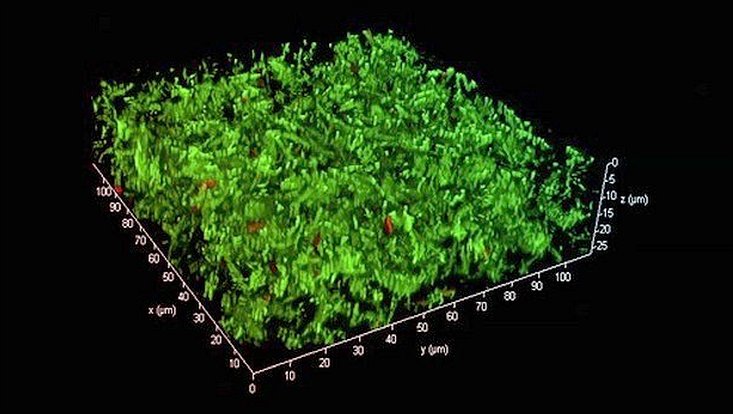
Foto: UHH/Mikrobiologie
3D-structure of a bacterial biofilm in a flow cell produced by Stenotrophomonas maltophilia K279a, an emerging pathogen (Cells were visualized with the life dead stain). The biofilms of Stenotrophomonas maltophilia clinical isolates were grown in flow cells under flow conditions at 28 °C for 72 hours. (Applied and Environmental Microbiology Vol. 86, No. 24, Alio et al., 2020)
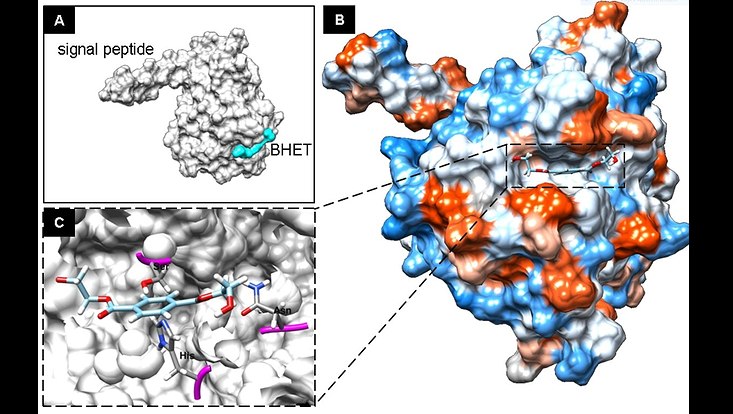
Foto: UHH/Mikrobiologie
3D-Structure prediction model of PET2 a metagenome-derived PET esterase and Bishydroxyethyl Therepthalic (BHET) acid fitting into the active site. The image was derived from Danso et al., 2018

Foto: UHH/Mikrobiologie
Cellulolytic bacteria forming colonizing cellulose fibres (unpublished image)

Foto: UHH/Mikrobiologie
Comamonas sp. forming biofilms on PET fibres. From the cover of AEM 19 and modified after Danso et al., 2019
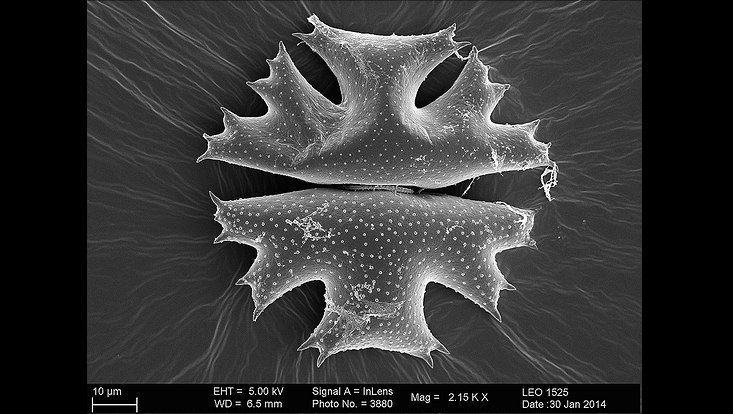
Foto: UHH/Mikrobiologie
Microbes colonizing microalga surfaces of Micrasterias crux melitensis. Small image shows a magnification of the core region of the microalga (unpublished).
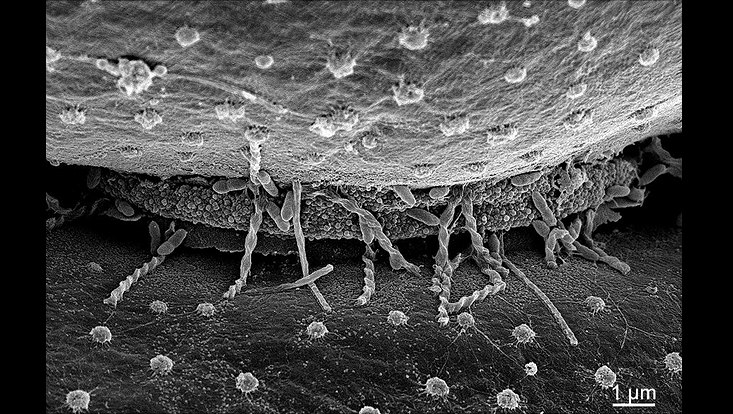
Foto: UHH/Mikrobiologie
A zoom into the center of the microalga Micrasterias crux melitensis

Foto: UHH/Mikrobiologie
Mining metagenomes for active hydrolases. Image derived from the cover of Green Chemistry issue 7, 2009 and Pottkämper et al., 2009
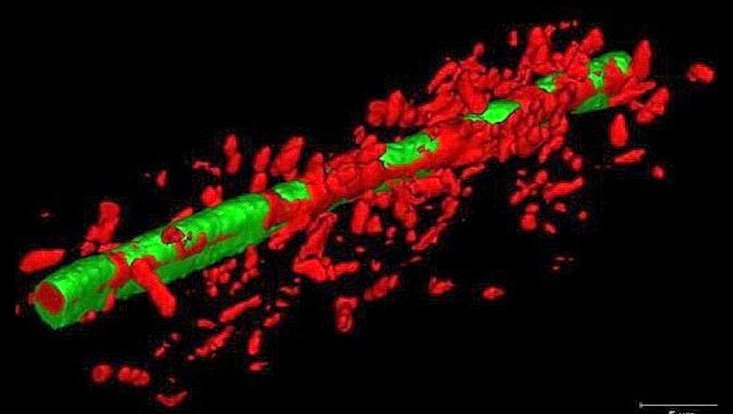
Foto: UHH/Mikrobiologie
Mikrobieller Biofilm von Janthinobacterium sp HH102 (rot) auf Filamenten des pflanzenpathogenen Pilzes Fusarium graminearum (grün) (Haack et al., 2016)
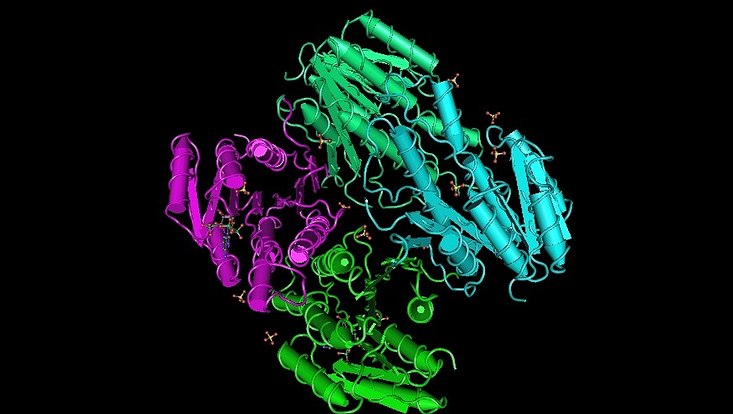
Foto: UHH/Mikrobiologie
3D-structure of a metagenome-derived oxidoreductase 3RKR involved in quenching auf Quorum sensing molecules in gram negative bacteria (Bijtenhoorn et al., 2011)
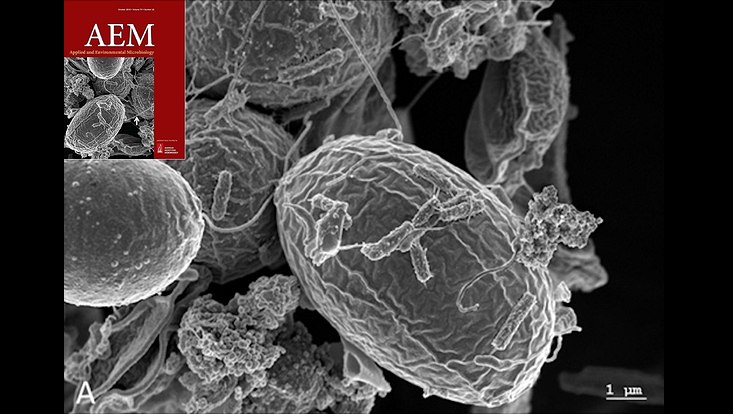
Foto: UHH/Mikrobiologie
Microbes colonizing microalga surfaces. The image was derived from Krohn et al.., 2013 and the cover image of AEM issue 79, (20) 2013.
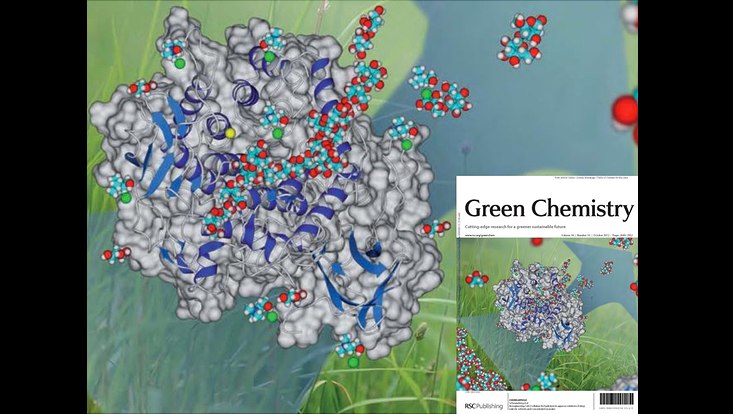
Foto: UHH/Mikrobiologie
3D-structure of the metagenome-derived and optimized cellulase CelA2 from the cover of Green Chemistry issue 10, 2012 and Lehmann et al., 2012
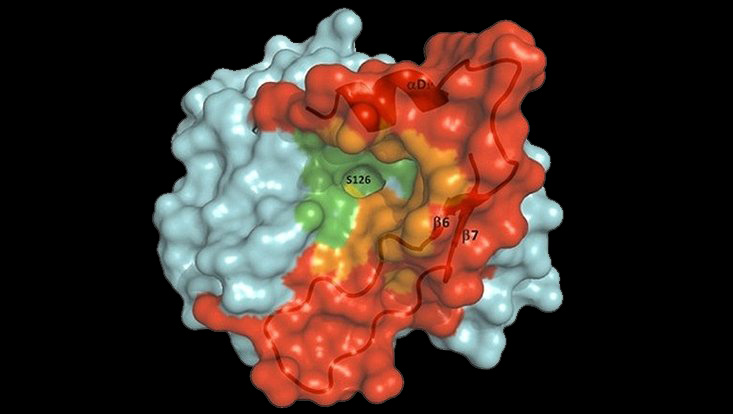
Foto: UHH/Mikrobiologie
3D-structure of the metagenome derived Lipase LipS (Chow et al., 2012, PDB: 4FBL). LipS is a true lipase derived from a non-cultivated Symbiobacterium sp. It has a high potential for application in biotechnology.
